分析技术研习室
课题组每周研讨会
基本概念
1.GEO:芯片数据的代表(动物)
- GSE—一个完整的研究,并提供了整个研究的描述,包括对数据的描述,总结分析。(series)
- GSM—用户提交给GEO的样本数据 (samples)
- GPL—用户测序使用的芯片/平台(platform)
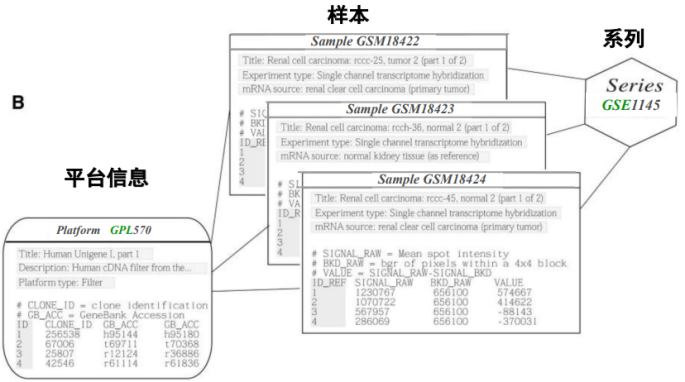
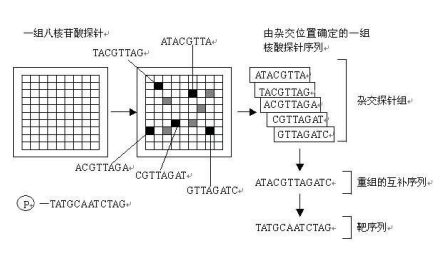
- 基因表达芯片
2.回归最初的问题:为什么要进行数据分析?
.png)
.png)
3.几个核心概念:
3.1 表达矩阵
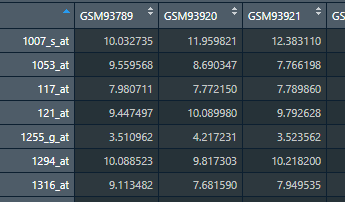
3.2差异分析
- 主成分分析(Principal Component Analysis,PCA): 作用:观察组别间的差异 主要概念:主成分—几个变量组成的 综合变量。
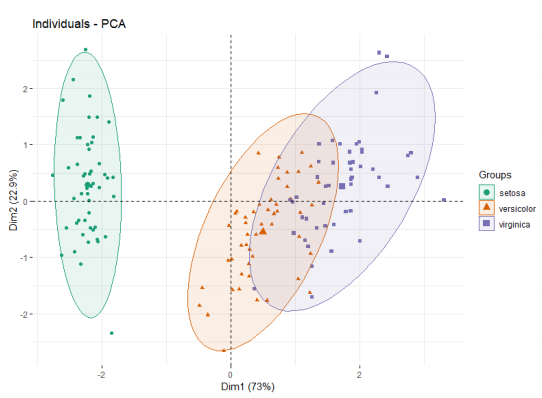
图中是利用PCA之后的聚类图。
Dim1~Dim2分别是指主成分1和主成分2,Dim1代表解释数据变化趋势最多的主成分,Dim2则是解释变化趋势第二多的主成分。 括号内的百分比则是代表了不同主成分所能解释数据变化趋势的比例。 中心位置的大点代表该组的位置。 一个点是一个sample,点距离代表相似性(差异)。
- limma包:本质上就是一个R包和里面的函数。
logFC&P.value
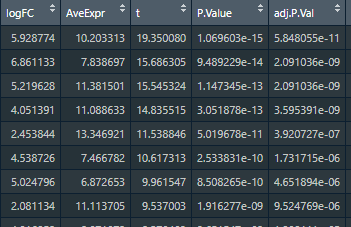
 log2(treat/control)=log2(treat)-log2(control)
log2(treat/control)=log2(treat)-log2(control)
.png)
通常所说的上、下调基因是指 基因表达量显著上、下调的基因:
上调基因:logFC>1, P.Value<0.01
下调基因:logFC<-1, P.Value<0.01
- volcano&heatmapP
3.3 富集分析
- GO
.png)
.png)
.png)
通过将差异基因做GO富集分析,可以把基因按照不同的功能进行归类,达到对基因 注释和分类 的目的。
- KEGG
.png)
数据分析思路
.png)
数据分析流程
.png)
- R包安装
options("repos"="http://mirrors.ustc.edu.cn/CRAN/")
if(!require("BiocManager")) install.packages("BiocManager",update = F,ask = F)
options(BioC_mirror="https://mirrors.ustc.edu.cn/bioc/")
cran_packages <- c('tidyr',
'tibble',
'dplyr',
'stringr',
'ggplot2',
'ggpubr',
'factoextra',
'FactoMineR',
'devtools')
Biocductor_packages <- c('GEOquery',
'hgu133plus2.db',
"KEGG.db",
"limma",
"impute",
"GSEABase",
"GSVA",
"clusterProfiler",
"org.Hs.eg.db",
"preprocessCore",
"hugene10sttranscriptcluster.db",
"enrichplot",
"ggplotify")
for (pkg in cran_packages){
if (! require(pkg,character.only=T) ) {
install.packages(pkg,ask = F,update = F)
require(pkg,character.only=T)
}
}
for (pkg in Biocductor_packages){
if (! require(pkg,character.only=T) ) {
BiocManager::install(pkg,ask = F,update = F)
require(pkg,character.only=T)
}
}
for (pkg in c(Biocductor_packages,cran_packages)){
require(pkg,character.only=T)
}
devtools::install_local("./AnnoProbe-master.zip",upgrade = F)
library(AnnoProbe)
- 致谢:生信技能树